Årsager og metoder til genomsekvensering
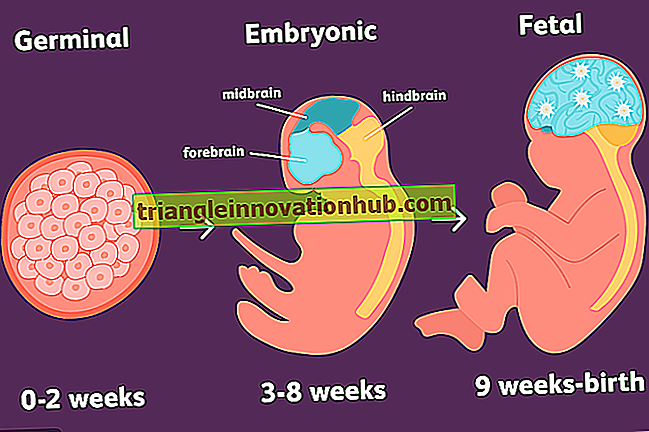
Sekventering af genomer er sofistikeret, kompleks og kræver specialiseret teknologi. Et segment på 500-800 bp kan sekventeres. Men genomerne er meget store, fx E. coli med 4, 2 x 10 6 bp og 3, 2 x 10 9 bp for mennesker.
Således er der behov for små segmenter til sekventering. Sådanne segmenter fremstilles ved at bryde genomisk DNA i små fragmenter tilfældigt. Sådanne fragmenter overlapper sædvanligvis andre fragmenter i deres ender. Fragmenter klones i en vektor for at danne et genomisk bibliotek af organisme.
Årsager til fuldstændig sekventering af et genom:
(i) Den tilvejebringer information som grundlag for opdagelsen af gener og giver opgørelse af gener.
(ii) Det repræsenterer de mulige forhold mellem generne.
(iii) Sekventering af genomer giver et sæt værktøjer til yderligere forsøg.
(iv) Genetisk information af en organisme stilles til rådighed fra gensekvens.
(v) Komplet genetisk sekventering af en organisme giver al genetisk information, der kræves for at danne en organisme.
Metoder anvendt til genomsekvensering:
(i) Direkte sekventering af Bacterial Artificial Chromosome (BAC) contigs:
Bakterielle kunstige kromosomer er simpelthen plasmider designet til kloning af meget lange segmenter (dvs. 100.000 til 300.000 bp) DNA. Disse vektorer bruges til at lave genomiske biblioteker, hvor indsatsstørrelsen er 80-100 kb. DNA-fragmenter af tusindvis af basepar klones ind i BAC-vektoren.
Det genomiske bibliotek screenes ved at lokalisere fælles restriktions kloner kaldet contigs. Medlemmerne af contig indeholder nogle overlappende regioner for at tillade præcis bestemmelse af deres placering i contig.
Det ultimative formål med fysiske kortlægningsmetoder er at modtage en komplet contig for hvert kromosom af genomet. De kortlagte contigs sekventeres ved at bryde store DNA-fragmenter i små segmenter. Hver klon af contig er sekventeret. Nukleotidsekvensen identificeres alene på klonbasis, indtil fuldstændigt genom er sekventeret.
(ii) Random shotgun sequencing eller bottom up approach:
Det er muligt at klone i en organisme et hvilket som helst ønsket gen fra en anden organisme ved skuddrivning. For dette hele genom af første organisme fordøjes med restriktionsendonuclease for at frembringe en tilfældig blanding af fragmenter.
Tilfældige (shotgim) biblioteker af genomisk DNA er konstrueret i små, dvs. 2, 0 kb og medium, dvs. 10 kb indsætter plasmidvektorer sammen med BAC-biblioteket med genomisk haglgevær (80-100 kb). Fragmenter indsættes i plasmidvektor, og de rekombinante plasmider transformeres til den ønskede værtscelle.
I skudpistol-sekventering sekventeres tilfældigt udvalgte kloner, indtil kloner i det genomiske bibliotek analyseres. Med andre ord kan vi sige, at i tilfældige skud pistol sekventering kromosomer er opdelt i sektioner og derefter sektionen af genomisk DNA er brudt i millioner af små segmenter og alle segmenterne er sekventeret på en upartisk måde.
Områder af overlappende DNA matches, hvilket dannede større og større segmenter af genomisk DNA. DNA-sekventering udføres i begge ender af indsatser af tilfældigt udvalgte kloner fra både de små og mellemliggende insert-plasmidbiblioteker for at tilvejebringe mindst tre gange dækning af genomet.

Små DNA-fragmenter indsættes tilfældigt i forskellige plasmidmolekyler. I tilfælde af overlappende indsatser kommer de alle fra samme genomiske placering, men fra forskellige regioner skiftes enten til venstre eller til højre i forhold til hinanden.
De overlappende fragmenter er indrettet enten til coting eller i en sekvens for det pågældende område (shot gun method). Prokaryotiske genomer har lidt respektive DNA- og skudpistol-sekventering, giver gode resultater. Eukaryoter bærer mange gentagne sekvenser.
Alt dette fører til forvirring. Overlappende fragmenter udnyttes imidlertid i samlingsfasen ved beregningsøvelse. Computerprogrammer identificerer de overlappende sekvenser og slutter dem til en kontinuerlig strækning. Denne proces er blevet anvendt succesfuldt til sekventering af alle mikrobielle genomsekvenser udgivet af Celera Genomics.